Лаборатория химии нуклеопротеидов, кафедра Химии природных соединений, Химический факультет
Публикаций
234
Цитирований
4 677
Индекс Хирша
38
Необходимо авторизоваться.
Лаборатория обладает широким спектром направлений исследований:
- Изучение рибосомы
- Поиск новых антибиотиков и изучение механизма их действия
- Изучение теломеразы
- Генетические основы канцерогенеза
- Поиск противораковых соединений
- Изучение аутофагии
- Поиск и изучение новых малых открытых рамок считывания
- Получение и изучение трансгенных клеточных линий и животных
- Структура белков методом ЯМР
- ЯМР-метаболомика
- CRISPR-Cas
- Вестерн-блот
- Культура клеток и тканей
- Toe-print
- ПЦР-РВ
- Работа с лабораторными животными
- Работа с бактериальными штаммами
- Клонирование
- Секвенирование по Сенгеру
- Выделение и очистка целевых белков
- In vitro трансляция
- Саузерн-блот
- Нозерн-блот
- Масс-спектрометрия
- Создание генноинженерных конструкций для экспрессии целевых белков
- Работа с прокариотическими клетками: культивирование, трансформация, работа с фагами
- Работа с эукариотическими клетками: культивирование, трансфекция, инфекция
- Выделение рибонуклеопротеидных комплексов, работа с рибосомами и их комплексами
- Аффинная хроматография
- Ультрацентрифугирование
- FISH (Флуоресцентная гибридизация in situ)
- Гель-электрофорез
Ольга Донцова
Заведующий лабораторией

Мария Зверева
Профессор
Мария Рубцова
Профессор
Петр Сергиев
Ведущий научный сотрудник
Радик Шафиков
Младший научный сотрудник

Екатерина Комарова
Младший научный сотрудник

Никита Шепелев
Младший научный сотрудник

Арина Никандрова
Стажёр-исследователь
Федор Грабовенко
Аспирант

Валерия Марина
Аспирант

Максим Кряквин
Студент

Анастасия Каракчиева
Студент
Виталий Буев
Студент

Юрий Карпенко
Студент
Святослав Балтин
Студент
Направления исследований
Первичное биотестирование de novo синтезированных химических препаратов
+
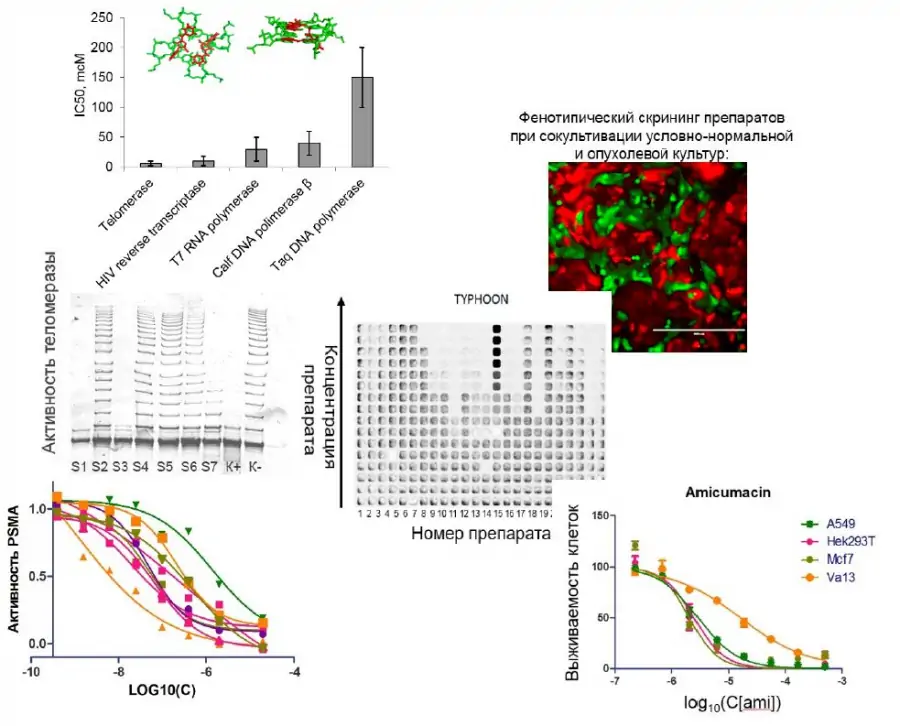
Данное направление подразумевает оценку цитотоксичности, в т.ч. дифференциальной, оценку непосредственного химического повреждения препаратами ДНК, адресный анализ предполагаемых мишеней препаратов (деблокирование в клетке белка p53, оценку ингибирования теломеразы, связывание с андрогеновыми рецепторами, ингибирование PSMA и др.). Работа ведется в тесном сотрудничеств с несколькими синтетическими группами на химическом факультете (на 3-м курсе студенты смогут сами “сварить” вещества в рамках курсовой работы по орг. химии и сами проверить их биологическое действие).
Изучение теломеразы
+
Изучение структурно-функциональных особенностей теломеразы с использованием в качестве модельной системы простейшего эукариотического организма – дрожжей Hansenula polymorpha. Термотолерантность этого вида дрожжей обеспечивает стабильность белков, что позволяет проводить структурные исследования компонентов теломеразы. Исследования теломеразы высших эукариот (в т.ч. изучение человека). Предметом особенного интереса является регуляция экспрессии основных компонентов теломеразы в человеческих клетках, биогенез теломеразной РНК и альтернативные функции фермента, не связанные с синтезом теломерной ДНК.
Поиск новых антибиотиков
+
Антибиотики подавляют рост клеток или приводят к их смерти. В отделе создана система высокопроизводительного скрининга потенциальных антибактериальных и цитотоксических препаратов, которая используется для анализа обширных коллекций соединений, как синтетического, так и природного происхождения. Репортная система, используемая в бактериях, позволяет на самом первом этапе скрининга выявить соединения, приводящие к ингибированию биосинтеза белка или ДНК. Коллекции соединений используются для поиска ингибиторов роста клеток эукариот. Скрининг токсичных против опухолевых клеток веществ позволяет выявлять соединения, преимущественно ингибирующие жизнедеятельность раковых клеток и не влияющие на выживаемость нормальных клеток в клеточной модели опухолевого микроокружения.
Изучение РНК-метилтрансфераз
+
Известно большое разнообразие различных дополнительных химических функциональных групп, которые присоединяются к нуклеотидам путем специфической модификации в клетке. Модифицированные основания в составе РНК играют важную роль в регуляции функционирования, структуры, как самих РНК, так и РНК-белковых комплексов. Многолетние исследования РНК-метилтрансфераз бактерий позволили выявить эволюционный аспект функционирования метилтрансфераз в клетках эукариот. Бионформатический анализ позволяет выявлять новые гены потенциальных РНК-метилтраснфераз, а последующие экспериментальные подходы идентифицировать ферменты и определять их мишени, а также влияние на функционирование клеток. В работе используются современные подходы, в том числе геномное редактирование при помощи системы CRISPR/Cas9 в линиях клеток эукариот и в лабораторных животных.
Публикации и патенты
Найдено
Ничего не найдено, попробуйте изменить настройки фильтра.
Головина А. Я., Дзама М. М., Остерман И. А., Сергиев П. В., Серебрякова М. В., Богданов А. А., Донцова О. А.
RU2522863,
2014
Остерман И. А., Андреянова Е. С., Сергиев П. В., Евфратов С. А., Донцова О. А., Иваненков Я. А., Лаптев И. Г., Плетнев Ф. И., Марусич Е. И., Леонов С. В., Головина А. Я.
RU2620074,
2017
Мажуга А. Г., Зверева М. Э., Агрон Л. А., Белоглазкина Е. К., Ворожцов Н. И., Донцова О. А., Зык Н. В., Киселев Ф. Л., Скворцов Д. А.
RU2468030,
2012
Новости лаборатории
Адрес лаборатории
Москва, Ленинские Горы, дом 1, стр. 40
Необходимо авторизоваться.



























